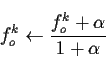
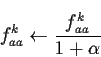
Alexandre de Brevern - Thèse de Bioinformatique Moléculaire
La méthode de prédiction bayésienne implique pour chaque bloc l'utilisation d'une matrice d'occurrence qui lui est propre. Ainsi, si un bloc est composé de deux types de séquences distinctes, l'utilisation d'une matrice commune entraîne un phénomène de moyennage qui fait perdre de l'informativité à ce bloc. Aussi pour améliorer la prédiction, nous avons mis au point la méthode "des familles séquentielles". Elle consite en la génération, pour un bloc protéique x de f matrices distinctes contenant chacune une partie des fragments protéiques du BP x. Pour cela, nous avons procédé à une classification des fragments par une méthode proche des cartes topologiques de Kohonen [109].
Pour un bloc protéique x, f matrices d'occurrence sont créées. Chacune des f matrices est initialisée en mettant les fréquences des acides aminés de la matrice associée au bloc x avec une légère variation pour les individualiser. Toutes les fréquences ont été recalculées pour avoir une fréquence égale à 1 en chaque position.
Ensuite, chaque fragment protéique associé au BP x est alloué à la matrice qui lui ressemble le plus parmi les f matrices. Pour cela, la probabilité conditionnelle Pl = P(XS/BPkl) est calculé pour l allant de 1 à f, avec XS la séquence en acide aminé correspondant au fragment. Ainsi, le score maximal Pl* = max {Pl} permet de caractériser la matrice l* correspondant au mieux au fragment XS. Cette matrice va être donc légèrement modifiée pour ressembler un peu plus au fragment XS. Chaque fréquence d'acides aminés faa en position k est modifiée.
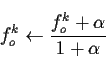
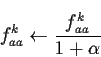
Cette transformation permet de conserver en chaque position une somme des fréquences toujours
égale à un. Le coefficient d'apprentissage ![]() est égal à
est égal à
![]() ,
avec
,
avec
![]() le taux initial d'apprentissage pris égal à 0,05, t représentant
le nombre de fragments déjà vus et Nk, le nombre total de fragments associés au bloc protéique x.
Le processus est itératif, l'ensemble des fragments est donc vu totalement à chaque cycle.
Au bout d'un certain nombre de cycles, les fragments se focalisent sur une seule des f matrices.
5 cycles ont été utilisés dans cette apprentissage.
le taux initial d'apprentissage pris égal à 0,05, t représentant
le nombre de fragments déjà vus et Nk, le nombre total de fragments associés au bloc protéique x.
Le processus est itératif, l'ensemble des fragments est donc vu totalement à chaque cycle.
Au bout d'un certain nombre de cycles, les fragments se focalisent sur une seule des f matrices.
5 cycles ont été utilisés dans cette apprentissage.
(c) 2001- Alexandre de Brevern