Alexandre de Brevern - Thèse de Bioinformatique Moléculaire
A chaque cycle, les fragments sont tirés aléatoirement. La figure 3.2 récapitule l'ensemble des deux étapes du processus d'apprentissage, la figure 3.3 pour la première et la figure 3.4 pour la seconde.
Dans un premier temps, l'apprentissage ne tient pas compte des transitions. (1) Un fragment est choisi aléatoirement dans la base de données d'apprentissage. (2) Son vecteur d'observation V(m) est comparé à chacun des blocs protéiques PBk à l'aide du critère du RMSda. B comparaisons sont donc calculées. (3) Le RMSda minimal correspond ainsi au bloc le plus proche structuralement du fragment. (4) Le vecteur W(k) du bloc le plus proche est très faiblement modifié pour ressembler à celui présenté au réseau V(m):
avec ![]() le coefficient d'apprentissage.
Le symbole
le coefficient d'apprentissage.
Le symbole
![]() veut dire "la valeur de gauche est remplacée par celle calculée à droite".
Ce coefficient, initialement pris faible, i.e.
veut dire "la valeur de gauche est remplacée par celle calculée à droite".
Ce coefficient, initialement pris faible, i.e. ![]() 0.02, décroit pendant l'apprentissage.
0.02, décroit pendant l'apprentissage.
![]() se modifie avec le nombre c de vecteurs d'observations déjà vus :
se modifie avec le nombre c de vecteurs d'observations déjà vus :
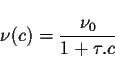
avec ![]() fixé arbitrairement à 1/N, N représentant
le nombre de vecteurs d'observations présents dans la
base de donnée d'apprentissage.
Dans notre cas, N = 56 442.
Ainsi,
fixé arbitrairement à 1/N, N représentant
le nombre de vecteurs d'observations présents dans la
base de donnée d'apprentissage.
Dans notre cas, N = 56 442.
Ainsi, ![]() est divisé par deux après le premier passage complet de la base.
est divisé par deux après le premier passage complet de la base.
L'apprentissage est itératif, un certain nombre de cycles C (= 15) est nécessaire pour définir des vecteurs optimaux W associés aux blocs. Chaque fragment n'est utilisé qu'une seule fois par cycle.
(c) 2001- Alexandre de Brevern