Alexandre de Brevern - Thèse de Bioinformatique Moléculaire
Prochain: Exemple
Au-dessus: Résultats
Précédent: Résultats
Sur l'ensemble des 342 structures protéiques recodées,
72 motifs de taille 5 ont été obtenus.
Leurs fréquences varient entre 17,3 % (14
729 observations) et 0,18 % (152 observations).
Les RMsd ont été calculés pour l'ensemble de ces motifs, appellés mots structuraux (les valeurs et la
description de ses mots sont disponibles sur le site web de l'Equipe de Bioinformatique Génomique et Moléculaire).
Les motifs les plus fréquents sont associés
aux formes des structures secondaires (mmmmm et ddddd).
Les 14 premiers ont servi à mettre en place le réseau et 44 des 58 suivants ont servi
le compléter.
La figure 5.1 montre le réseau obtenu.
Ce réseau est ainsi composé de
31 n uds et utilise 15 des 16 BPs.
Les couleurs représentent les occurrences observées, ainsi
l'entrée principale en hélice
uds et utilise 15 des 16 BPs.
Les couleurs représentent les occurrences observées, ainsi
l'entrée principale en hélice 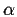 fkl (n
fkl (n uds 08-09-10)
vers le BP m (n
uds 08-09-10)
vers le BP m (n ud 01) a une
sortie principale nop (n
ud 01) a une
sortie principale nop (n uds 02-03-04) et deux
sorties plus courtes (et moins observées) pcc (n
uds 02-03-04) et deux
sorties plus courtes (et moins observées) pcc (n uds 20-21-22) et c (23).
La partie entourant le site du BP d (n
uds 20-21-22) et c (23).
La partie entourant le site du BP d (n ud 07) est plus
complexe, l'entrée s'effectue par un seul n
ud 07) est plus
complexe, l'entrée s'effectue par un seul n ud c (n
ud c (n ud 06),
celui-ci étant compris dans une succession nopac (n
ud 06),
celui-ci étant compris dans une succession nopac (n uds 02-03-04-05-06) ou
kbc (n
uds 02-03-04-05-06) ou
kbc (n uds 31-27-06),
sa sortie la plus importante est la série dehiacd (n
uds 31-27-06),
sa sortie la plus importante est la série dehiacd (n uds 07-25-16-17-18-19-07).
La seconde sortie du feuillet la plus
importante est la succession dfkl (n
uds 07-25-16-17-18-19-07).
La seconde sortie du feuillet la plus
importante est la succession dfkl (n uds 07-08-09-10).
Ensuite la sortie plus complexe de df (n
uds 07-08-09-10).
Ensuite la sortie plus complexe de df (n uds 07-13) allant vers les
sites b (n
uds 07-13) allant vers les
sites b (n ud 14) et k (n
ud 14) et k (n ud 31).
ud 31).
![\begin{sidewaysfigure}% latex2html id marker 2616
[htbp]
\centerline{\epsfxsize...
...des terminaisons du graphe (voir figure \ref{fig_graph2}).}
\end{sidewaysfigure}](Pictures/img192.gif)
Des parties moins observées ont était mises comme le dédoublement du BP e (n ud 26-15) en amont du site
h (n
ud 26-15) en amont du site
h (n ud 16), la sortie en j
(n
ud 16), la sortie en j
(n ud 25) de ce dernier, la suite alternative de fkl (n
ud 25) de ce dernier, la suite alternative de fkl (n uds 08-09-10)
vers pc (n
uds 08-09-10)
vers pc (n uds 11-12) ou les deux passages de dfk (n
uds 11-12) ou les deux passages de dfk (n uds 07-13-31)
vers bc (n
uds 07-13-31)
vers bc (n uds 27-06) ou op (n
uds 27-06) ou op (n uds 03-04).
Le réseau final obtenu est visible dans un plan 2D sans aucune intersection des flèches.
Le caractère discontinu du réseau doit être noté, ainsi tous les
fragments conservés sont ceux
qui peuvent s'inscrire dans le réseau,
c'est à dire s'ils ont 5 sites consécutifs.
La figure 5.2 donne les fréquences de début et de fin
des motifs observés sur le réseau, cad
la fréquence relative de chaque n
uds 03-04).
Le réseau final obtenu est visible dans un plan 2D sans aucune intersection des flèches.
Le caractère discontinu du réseau doit être noté, ainsi tous les
fragments conservés sont ceux
qui peuvent s'inscrire dans le réseau,
c'est à dire s'ils ont 5 sites consécutifs.
La figure 5.2 donne les fréquences de début et de fin
des motifs observés sur le réseau, cad
la fréquence relative de chaque n ud pour être le premier
ou le dernier résidu d'un fragment protéique compris dans le graphe.
Seul le site c (n
ud pour être le premier
ou le dernier résidu d'un fragment protéique compris dans le graphe.
Seul le site c (n ud 06) situé
juste avant le bloc d (feuillet
ud 06) situé
juste avant le bloc d (feuillet  , n
, n ud 07) possède des taux largement supérieurs
aux autres sites.
Ce fait s'explique à la fois par des différences de tailles et de formes
de structures associées aux feuillets
ud 07) possède des taux largement supérieurs
aux autres sites.
Ce fait s'explique à la fois par des différences de tailles et de formes
de structures associées aux feuillets  et surtout
au principe même du graphe
où 3 des 4 terminaisons sont des n
et surtout
au principe même du graphe
où 3 des 4 terminaisons sont des n uds correspondant au PB c:
en fin d'hélice
uds correspondant au PB c:
en fin d'hélice 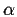 n
n uds 22 et 23 des séries mc et mpcc
(en haut à gauche de la figure 5.1)
et le n
uds 22 et 23 des séries mc et mpcc
(en haut à gauche de la figure 5.1)
et le n ud 12 de la série lpc (en bas du graphe).
ud 12 de la série lpc (en bas du graphe).
![\begin{sidewaysfigure}% latex2html id marker 2653
[htbp]
\centerline{\epsfxsize...
...es de fin juste en-dessous
des pr\'ec\'edentes en bleues.}
\end{sidewaysfigure}](Pictures/img193.gif)



Prochain: Exemple
Au-dessus: Résultats
Précédent: Résultats
Page 99
(c) 2001- Alexandre de Brevern